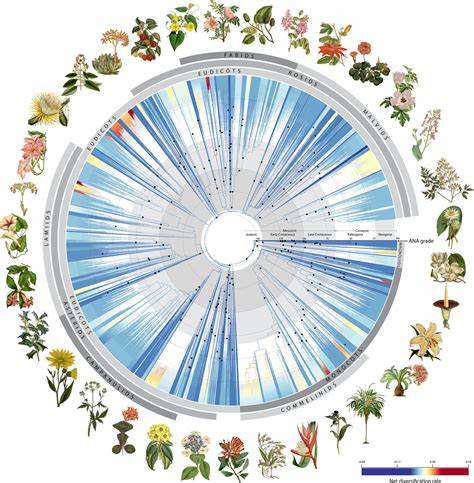
В современном мире биология и экология всё чаще используют генетические методы для изучения биоразнообразия, исследуя огромное количество видов и их эволюционные связи. Традиционный подход к идентификации видов — ДНК-баркодирование — за последние два десятилетия доказал свою пользу в самых разных сферах науки и практики, от эволюционных исследований до охраны окружающей среды. Однако, несмотря на широкое применение, классические методы ДНК-баркодирования имеют определённые ограничения, такие как недоступность универсального маркера для всех организмов, трудности в дифференциации близкородственных видов и значительные затраты времени и ресурсов при анализе большого массива геномных данных. В связи с этим новая концепция универсальной ДНК-подписи, созданная методом varKoding, открывает невероятные возможности для научного сообщества и практических приложений во всем мире. Метод varKoding построен на идее использования очень низкозатратного геномного сканирования (геномный скиминг) с минимальным покрытием, что позволяет получать обобщённые и статистически значимые данные о составе нуклеотидов в ДНК, не требуя полной сборки генома.
Основная инновация заключается в создании двумерного изображения — так называемого varKode — на основе распределения коротких последовательностей ДНК (k-миров) и их частот. Эти изображения выступают своего рода геномной подписью, уникальной для каждого вида или таксона и отображающей особенности его геномного состава. Созданные таким образом varKodes используются в сочетании с мощными алгоритмами машинного обучения, в частности нейронными сетями, которые, обучаясь на множестве таких «изображений», способны быстро и точно классифицировать новые образцы по представленным образцам. Интересно, что технология опирается на современные архитектуры, такие как трансформеры (Vision Transformers, ViT), что обеспечивает высокую точность даже при очень небольшом объеме входных данных (от 500 тысяч до нескольких миллионов базовых пар), что существенно сокращает время и стоимость секвенирования. Ещё одним важным достижением является универсальность метода.
В отличие от классических подходов, где для каждого царства живых организмов (растения, животные, грибы и пр.) применяются свои специализированные маркеры и методики, varKoding применима ко всему дереву жизни: от бактерий и архей до цветковых растений, грибов и животных. Проведённые эксперименты и проверки охватывали обширный спектр таксонов — как внутривидовые, так и межвидовые различия — демонстрируя стабильную высокую точность — выше 90% в различных сценариях. Метод varKoding выгодно отличается также и по части необходимости в начальных данных. Благодаря способности извлекать информативные паттерны из низкозатратных геномных данных, технология открывает доступ к изучению образцов с ограниченным качеством ДНК, таким как гербарии, музейные коллекции, а также краеведческие и экологические исследования, где высокий объем препаратов получить сложно или дорого.
Учитывая возрастающее количество доступных геномных данных и постоянное улучшение технологий секвенирования, варианты применения varKoding становятся практически безграничными. Одно из ключевых преимуществ метода — масштабируемость. В то время как альтернативные методы, основанные на полных геномах и парных сравнениях между образцами, требуют экспоненциального увеличения вычислительных ресурсов с ростом объема данных, в varKoding нейронная сеть имеет фиксированную архитектуру и фокусируется на обучении и распознавании характеристик, что делает процесс менее затратным и гораздо более быстрым. Более того, сохранённые varKode-изображения занимают весьма небольшой объем памяти, что облегчает их хранение и передачу. Потенциал varKoding выходит далеко за пределы академических исследований, затрагивая множество отраслей.
Например, экологический мониторинг и выявление инвазивных видов могут получить существенный прирост эффективности за счёт быстрого анализа окружающей среды с минимальными требованиями к образцам. Медицинские и судебно-экспертные учреждения смогут более точно идентифицировать патогенные микроорганизмы или устанавливать происхождение биологических образцов, при этом затрачивая меньше времени и средств. Также varKoding представляет интерес для фармакологии и сельского хозяйства: оценка сырья, прослеживание происхождения, борьба с контрафактной продукцией и биобезопасность выйдут на новый уровень качества. Исследования, связанные с varKoding, предоставляют также уникальный взгляд на эволюционные изменения в геномах. Изучение распределений коротких последовательностей и их частот, отражённых в varKode, помогает выявлять закономерности, связанные с адаптацией, гибридизацией, трансгенными событиями и даже повреждениями ДНК.
Таким образом, метод способствует не только идентификации, но и фундаментальному пониманию механизмов биоразнообразия. Необходимо отметить и перспективы дальнейшего развития. Благодаря мультидисциплинарному подходу, который объединяет биоинформатику, компьютерное зрение и молекулярную биологию, инструментарием varKoding можно дополнительно расширять. Новые алгоритмы глубокого обучения и усовершенствованные способы представления геномных данных позволят повысить универсальность и чувствительность, а также упростят работу с комплексными образцами, такими как метагеномы и смешанные биологические сообщества. Важным направлением также является оптимизация и автоматизация подготовки данных.
Создание портативных устройств и автоматизированных лабораторных систем на основе nanopore-секвенирования с возможностью прямого получения varKode изображений позволит проводить идентификацию непосредственно в полевых условиях или при ограниченных ресурсах. На сегодняшний день varKoding развивается в рамках открытых проектов, доступных для широкой научной общественности, что стимулирует обмен данными и совместные усилия по созданию богатых эталонных баз и улучшению алгоритмов. Такой подход способствует ускорению внедрения методики в практические задачи и обеспечивает прозрачность результатов. Резюмируя, технология композитной универсальной ДНК-подписи через varKoding — это современный прорыв в изучении и мониторинге биоразнообразия. Она удачно сочетает в себе универсальность, точность, экономичность и масштабируемость, позволяя решать задачи, недоступные прежним методам.
Благодаря этой инновации в ближайшем будущем биологи, экологи и специалисты различных отраслей смогут более эффективно и быстро изучать мир живых организмов, раскрывая тайны эволюции и взаимодействия природы.