Современная биоинформатика требует мощных и при этом удобных инструментов для анализа множественных выравниваний последовательностей (МСА), что особенно важно для изучения структуры и функции белков, РНК и других биомолекул. Одним из таких инструментов является Evorca - быстрое и минималистичное программное обеспечение, реализующее подход plmDCA (псевдо-логарифмическую максимизацию потока данных) на базе библиотеки JAX. Его высокая производительность, простота в использовании и гибкость делают Evorca привлекательным выбором как для исследователей, так и для разработчиков новых биоинформационных приложений. Evorca специализируется на оценке статистических парных взаимодействий внутри МСА посредством обучения модели Поттса, что позволяет выявлять контактные карты и матрицы взаимодействия аминокислот или нуклеотидов. В основе лежит минимизация отрицательного псевдо-логарифмического функционала при регуляризации параметров, что гарантирует устойчивость и точность оценки сложных взаимосвязей в данных выравниваний.
Инструмент обрабатывает данные с помощью эффективных вычислений на основе JAX и Optax, обеспечивая поддержку как процессоров, так и ускорителей GPU. Основным преимуществом Evorca является его компактность и простота - архитектура кода минималистична и понятна, что облегчает понимание методологии и расширение функционала под конкретные задачи. Пользователи могут работать с программой через удобный интерфейс командной строки, ускоряя запуск расчетов, или интегрировать в более сложные пайплайны с помощью Python API, ориентированного на работу с массивами NumPy, что заметно повышает продуктивность анализа. Процесс работы Evorca начинается с преобразования входного МСА формата A3M: удаляются вставки, кодируются последовательности с использованием специфичных алфавитов (включая 20 аминокислот для белков или ACGU для РНК), а также выполняется подсчет весов последовательностей по схеме, учитывающей уменьшение избыточности при игнорировании пробелов. Далее строится модель с двумя наборами параметров - одиночные "поля" для каждого положения и парные взаимодействия между парами позиций.
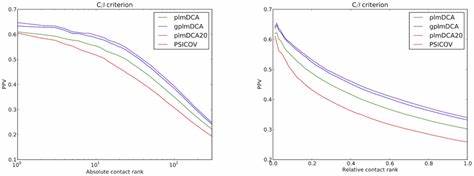
Оптимизация выполняется с помощью алгоритма AdamW, адаптированного для использования через Optax, что ускоряет сходимость и повышает стабильность результата. Особое внимание уделено корректному обращению с симметрией парных взаимодействий и исключению самосвязей, а также применению "нулевого суммирования" по каждой паре позиций для выравнивания модели. Итогом является получение матриц взаимодействия, по которым вычисляется скалярный показатель - норма Фробениуса - отражающий силу и значимость связи между позицией в белке или РНК. Для повышения качества предсказаний применяется коррекция методом среднепродуктовой коррекции (APC), исключающая артефакты из-за общей консервативности регионов. Результаты работы Evorca включают несколько форматов выходных данных.
Например, h.npy содержит параметры одиночных состояний, sparse_J.npz - разреженные матрицы парных взаимодействий с сохранением верхнетреугольной структуры, а Jsym.npy представляет симметризованные и откалиброванные матрицы параметров. Кроме того, score.
npy сохраняет итоговые значения норм Фробениуса, а contact_map.png визуализирует эти данные в форме контактной карты, полезной для быстрого анализа и интерпретации. Для пользователей предусмотрены удобные сценарии запуска. Через CLI можно за несколько команд подготовить модель, провести обучение и сгенерировать визуализации, что идеально подходит для оперативного анализа больших МСА. Для интеграции в научные исследования предлагается Python API с поддержкой гибких параметров, позволяющий непосредственно работать с массивами данных без необходимости записи файлов - это удобно для последующего анализа и автоматизации.
Отдельно стоит отметить, что Evorca ориентирован на эффективность при работе с большими выравниваниями благодаря использованию разреженного ввода-вывода и вычислительных оптимизаций JAX, что позволяет существенно снизить требования к ресурсам и повысить скорость работы без жертвования точностью. Возможность запускать программы на GPU с помощью соответствующих сборок JAX дополнительно усиливает производительность и сокращает время обучения. Evorca занимает важное место среди инструментов для анализа множественных выравниваний, подчеркивая важность баланса между скоростью, точностью и простотой использования. Благодаря открытому исходному коду и лицензии MIT, программисты и ученые могут адаптировать инструмент под свои нужды и внедрять его в разнообразные исследовательские рабочие процессы, ускоряя выявление структуры белков и функциональных взаимодействий. Среди областей применения Evorca выделяются структурная биоинформатика, молекулярная биология и биофизика, где точное и быстрое определение контактных карт позволяет улучшать модели 3D-структур белков и РНК, быстро выявлять важные функциональные участки и потенциально упростить дизайн лекарственных препаратов.
Поскольку анализ делается на основе статистических взаимосвязей в большом числе гомологических последовательностей, эти методы отлично дополняют экспериментальные подходы и решают задачи, недоступные традиционным методам. Работа с MSA формата A3M предполагает удаление вставок и стандартизированное кодирование последовательностей, что позволяет сделать моделирование более корректным и уменьшает шум в данных. Алгоритмическое выполнение взвешивания последовательностей согласно схемам, предлагаемым Хэникoffом, дополнительно снижает избыточность, делая результаты более надежными и интерпретируемыми. Псевдологарифмический подход к обучению модели Поттса, который реализован в Evorca, отличается от классического максимального правдоподобия тем, что оптимизируется условная вероятность одной позиции при фиксированных остальных, что значительно упрощает расчеты и повышает масштабируемость. Это критичное преимущество, позволяющее эффективно обрабатывать реальные биологические данные с многочисленными позициями и состояниями.
Особенность выбора JAX в качестве базовой платформы для вычислений заключается в том, что этот фреймворк сочетает в себе динамическую автодифференциацию, поддержку GPU и TPU, а также интеграцию с популярными оптимизаторами через Optax, что обеспечивает как гибкость разработки, так и высочайшую производительность. Таким образом, Evorca представляет собой современный, легкий и высокопроизводительный инструмент, оптимально подходящий для решения задач анализа множественных выравниваний и поиска контактных взаимодействий в биомолекулах. Он обеспечивает качественную базу для дальнейших исследований, интеграции в вычислительные пайплайны и стимулирует развитие методов статистического моделирования в биоинформатике. Пользователи, стремящиеся к эффективному и простому решению задач plmDCA, найдут в Evorca сочетание производительности, удобства и точности, что делает этот проект перспективным и актуальным выбором для научного сообщества по всему миру. .

![A Technical Analysis on the Chinese Great Firewall [pdf]](/images/EFEA12A3-42E5-4773-8DEB-901FC6DAC302)