Идентификация биологических видов с помощью ДНК-баркодирования стала одним из ключевых методов в современной биологии и экологии, открывая широкие возможности для изучения биоразнообразия, мониторинга экосистем и биомедицинских применений. Тем не менее, традиционные методы ДНК-баркодирования обладают рядом ограничений, мешающих их применению на всех масштабах и для всего многообразия живых организмов. Специализированные баркоды, зависящие от конкретных групп организмов, низкая разрешающая способность для близкородственных видов, проблемы с мутациями, гибридизацией и качество исходного материала вносят значительные преграды. Более того, методы, основанные на полной последовательности геномов, хотя и способны преодолеть многие ограничения, слишком требуют больших ресурсов и сложно масштабируемы для миллионов существующих видов. В этих условиях появление метода varKoding, применяющего универсальную ДНК-подпись, становится знаменательным событием в эволюционной биологии и биоразнообразии.
ВарКодинг — это инновационный метод, который использует низкоковерные геномные данные (так называемый «genome skim») для создания двухмерных изображений, в которых отражены частоты коротких последовательностей ДНК (k-меров) и их вариации в геноме каждого вида. Эти изображения называются varKodes и служат своеобразным графическим «отпечатком» геномной структуры. Применение нейронных сетей, в частности архитектур визуальных трансформеров (ViT), обеспечивает высокоточное распознавание таких подписи даже при минимальных объёмах данных. Таким образом, varKoding объединяет экономичность получения данных и современные алгоритмы машинного обучения, чтобы получить универсальный и масштабируемый инструмент идентификации. Особенность этого подхода заключается в том, что требуемое количество данных для точной идентификации вида существенно ниже, чем у других методов — порядка 10 мегабаз последовательностей и менее.
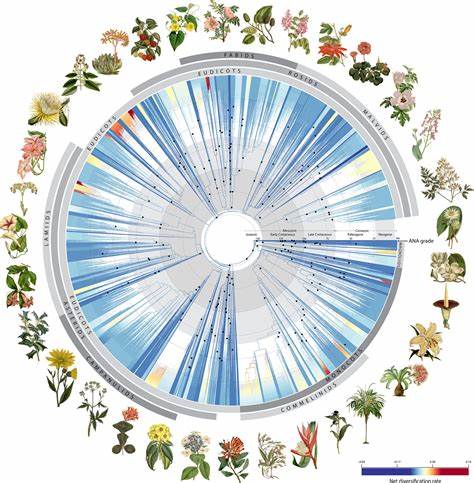
Это возможно благодаря умелому использованию статистики распределения k-меров с ранжированной трансформацией, позволяющей уменьшить влияние артефактов и загрязнителей в данных. Помимо экономии затрат на секвенирование, метод отличается высокой устойчивостью к разнообразию источников ДНК, включая образцы с повреждённой или деградированной матрицей, что важно для работы с музеальными коллекциями и древними образцами. Разработка и апробация varKoding велись на широком наборе таксонов, охватывающем цветковые растения (семейство Malpighiales), животных (жуки Bembidion), грибов (лихенообразующие грибы Xanthoparmelia) и даже бактерий (Mycobacterium tuberculosis). Во всех случаях было продемонстрировано превосходство по точности, воспроизводимости и мощности над существующими методами — от классического ДНК-баркодирования и фрагментного секвенирования до алгоритмов анализа ортологии и поиска гомологии. Важное достоинство varKoding — это универсальность: один и тот же алгоритм, основанный на обработке varKodes, применим для идентификации организмов на самых разных уровнях таксономии — от вида до семейства и выше, вплоть до границ царств жизни.
Такая масштабируемость и стандартизация позволяют создавать единые базы данных и обширные справочные модели, которые значительно облегчают глобальные проекты по каталогизации биологического разнообразия. При работе с большими базами данных, содержащими сотни тысяч и миллионы геномных образцов, varKoding показывает высокую вычислительную эффективность: время обработки растёт линейно, а не квадратично, как это происходит у методов сравнения попарных расстояний. Это, в сочетании с относительной лёгкостью получения и хранения varKode-изображений (несколько килобайт вместо сотен мегабайт данных), делает технологию перспективным решением для проектов, таких как Earth BioGenome Project и других инициатив по секвенированию всего живого на планете. Кроме того, varKoding открывает новые возможности для приложений в области судебной генетики, мониторинга окружающей среды, системы идентификации товаров и ресурсов, борьбы с незаконной торговлей редкими видами и даже исследования древней ДНК. Возможность идентифицировать образцы с минимальным секвенированием даёт шанс проводить быстрый и экономичный анализ в полевых условиях при помощи портативных секвенаторов, таких как Oxford Nanopore.
Методика не требует сложного сбора и подготовки данных и может быть адаптирована для получения таксономической, географической или иной информации о происхождении образца. Научное сообщество уже получило доступ к предварительно обученным моделям varKoding, размещённым на платформах Huggingface, что способствует быстрому тестированию и распространению методов среди исследователей без необходимости облачных вычислительных ресурсов. Несмотря на многообещающие результаты, разработчики отмечают ряд перспективных направлений для дальнейших исследований и улучшений. Например, исследование пределов работы технологии с сильно деградированной ДНК, а также возможности обработки смешанных образцов, характерных для метагеномных исследований. Кроме того, углублённый анализ тех геномных признаков, которые наиболее значимы для дифференциации видов с помощью varKoding, поможет лучше понять эволюционные процессы и особенности геномной структуры различных таксонов.